来源:晶泰科技
研究摘要
疫情出现后,晶泰科技于1月20日迅速成立研究小组,调集云端的巨大算力,对病毒感染宿主机理提供分子层面的研究,为病毒预警和防治、及药物开发提供基础研究和数据开源支持。
通过计算出Spike蛋白与ACE2配体的结合自由能和构象,在一定程度上预测验证了病毒较大的人际传播可能性
基于已公布的研究,在新型冠状病毒感染人体的过程中,Spike蛋白很可能是与人体内ACE2配体结合。通过高精度计算这对蛋白的结合自由能和构象,可以从分子层面理解病毒感染机理。我们的高精度计算结果显示,2019-nCoV病毒与人体ACE2蛋白的亲和力可能略高于SARS。这一计算在一定程度上预测验证了病毒较大的人际传播可能性。基于该计算方法,判定未来同类病毒的人际传播风险的分子层面量化模型评估或可在一天内完成,帮助一线研究者快速、多维预测疾病威胁。
利用高精度的自由能微扰(XFEP)方法,对Spike蛋白与人体ACE2配体结合位点的蛋白质关键残基进行了分析和丙氨酸扫描,从而预测病毒及其突变对传染性和致病性的影响
2019-nCoV属于RNA病毒,在其自我复制的传播过程中,病毒表面的S蛋白会较快发生突变(目前已经出现了含有S蛋白突变的变异毒株【3】)。通过高精度计算,可以获知有哪些病毒上氨基酸的突变可能增强病毒与人体内特定蛋白结合的能力。这一计算结果,可以预判可能出现的感染力更强的突变毒株,提前开发相应的多肽或抗体类药物,快速应对病毒变异,从而减少潜在的风险和损失。
针对全球已上市药物小分子及重要分子的老药新用筛选,找到183个对病毒有潜在治疗效果的药物,并给出活性排序
药物研发过程漫长,从已上市的药物分子中筛选出对病毒有抑制效果的药物可以省去大部分毒副作用等安全性试验与药剂合成、开发、投入生产的时间,从而大大缩短找到有效药物并开始投入使用的时间。晶泰科技利用2019-nCoV病毒的几个关键蛋白的三维结构模型,从数千种上市药与一万种中药成分中筛选出有可能有效抑制新型冠状病毒的183个药物分子,扩大了筛选老药的寻找范围,为尽快推进动物、临床试验,找到具有活性的治疗方案打下基础。
目前,研发小组目前正在针对Remdesivir (GS-5734)等高关注度药物完成活性重点分析,并继续完成病毒Spike蛋白的丙氨酸扫描,综合评估预测病毒变异对其传染性、致病性的影响。
现将阶段性研究成果与计算数据与同行共享,希望我们的工作能为战斗在抗击疫情一线的科学家和医务工作者们提供帮助,加快找到有效药物的进程,为疫苗研发与临床治疗提供科学参考与研究基础。
晶泰科技也在积极与武汉的实验科研组建立细胞层面试验数据的信息通路,同时寻找合作进行动物实验,用于进一步确认小分子单药和联合用药治疗效果。希望有实验验证能力及合作意愿的企业和研究团队与我们联系,协力同心,攻坚克难,共同加速2019-nCoV的治疗药物研发。
点击“阅读原文”,获取以下内容:
2019-nCoV病毒Spike蛋白同源建模模型结构
2019-nCoV病毒Spike蛋白MD优化后的模型结构
基于XFEP的丙氨酸扫描结果
2019-nCoV 病毒3CLpro和PLpro潜在靶点的同源建模蛋白结构
致信ncov@xtalpi.com,获取:
183个对3CLpro和PLpro靶点具备潜在抑制作用的上市药及中药成分列表及活性排序
1.Spike蛋白结合构象及亲和力研究
2019-nCoV病毒表面的S蛋白(Spike Glycoprotein, 刺突糖蛋白)在病毒感染中负责结合宿主受体细胞,并促进病毒与细胞膜的融合,是小分子、多肽药物开发的潜在靶点。最新研究显示,S蛋白在人体中结合的受体蛋白很可能是ACE2蛋白(Angiotensin-Converting Enzyme 2,受体血管紧张素转化酶2)【1】。
以SARS病毒S蛋白在结合状态下的RBD区域作为模版(PDB ID: 2AJF,【2】),我们对新型冠状病毒S蛋白S1亚基的受体结合区(Receptor Binding Domain,RBD) 与ACE2所形成的复合物进行了同源建模(图1)和分子动力学模拟,以预测S蛋白的RBD在结合ACE2以后可能发生的构象变化。
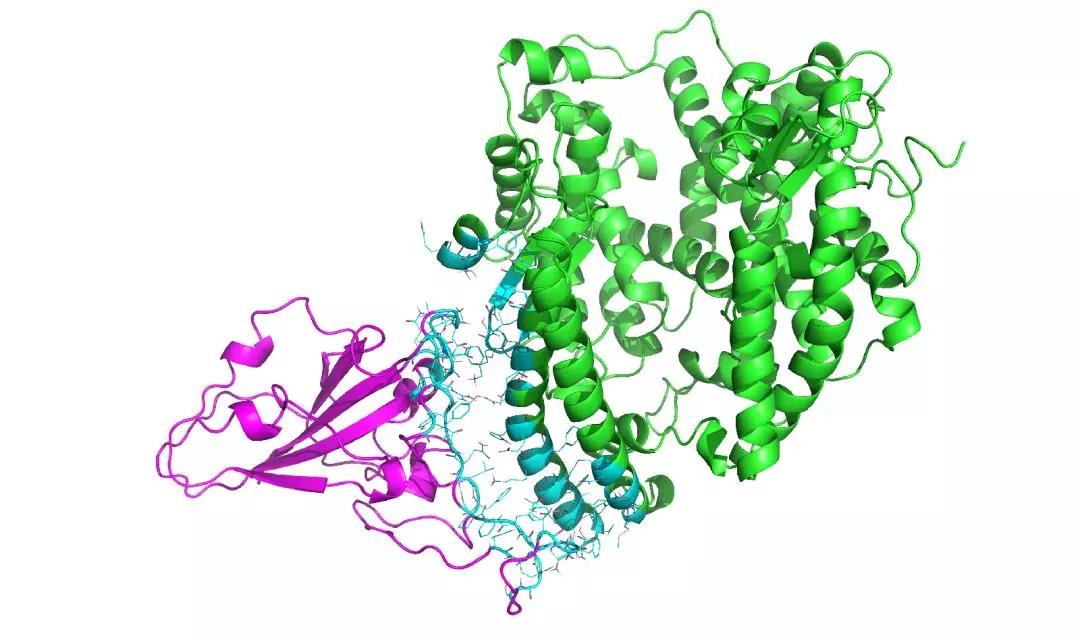
图1:通过同源建模得到的2019-nCoV S蛋白与受体ACE2蛋白的结合模式。其中粉色的是2019-nCoV S蛋白,绿色的是ACE2,蓝色的是两个蛋白中有相互作用的残基。
S蛋白RBD和ACE2形成复合物的构象优化
晶泰科技新冠状病毒研究小组对2019-nCoV RBD-ACE2复合物进行了100 纳秒的分子动力学模拟,在平衡后的动力学模拟过程中, 未对受体和病毒蛋白质的坐标进行任何约束。
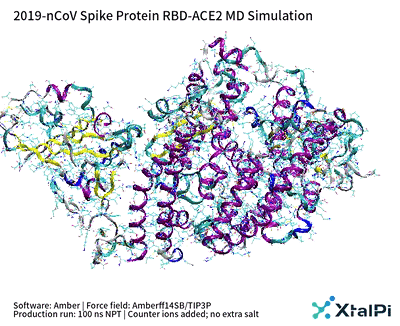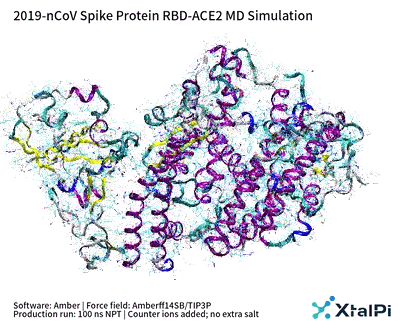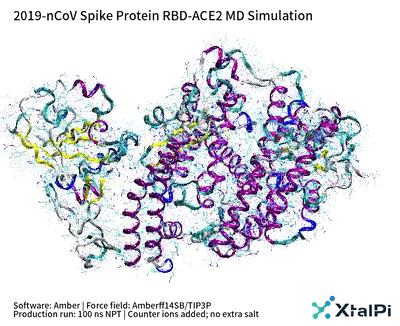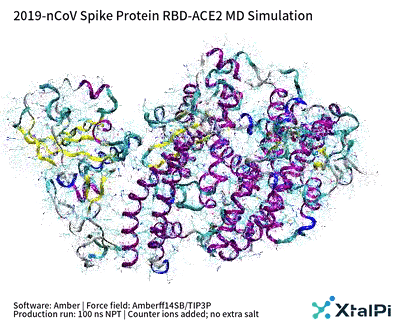
图2:2019-nCoV Spike 蛋白 RBD-ACE2 MD 模拟
模拟的轨迹显示,复合物的结构在模拟的过程中始终维持相对稳定,并在结合界面处形成了大量的氢键结构和疏水相互作用。初始的复合物的结构和轨迹最后一帧的坐标之间的RMSD为1.89 Å (图3)。在此基础上,采用晶泰科技的XFEP高精度自由能微扰(Free Energy Perturbation, FEP)方法,对受体结合区的关键氨基酸进行丙氨酸扫描 (Alanine Scan),进而得到S蛋白可能的突变对病毒入侵人体细胞过程产生的影响。
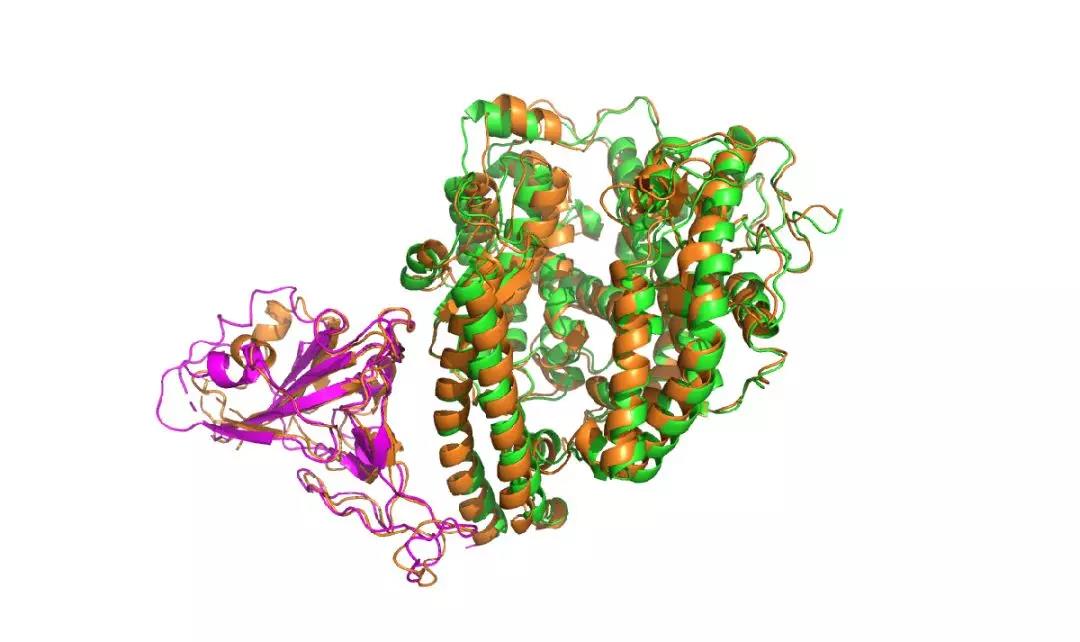
图3:复合物2019-nCoV RBD-ACE2 的初始结构(粉色、蓝色)和100 纳秒分子动力学模拟轨迹的最后一帧(橙色)结构的叠合
基于XFEP方法的丙氨酸扫描
冠状病毒通过S蛋白S1亚基上的RBD与ACE2受体结合来感染人体细胞,因此通过计算S蛋白不同突变体与ACE2的结合力变化,可以在一定程度上预测病毒突变体的传染性和致病性变化。计算和观察发现,2019-nCoV病毒在受体结合界面上相对于SARS-CoV多处残基类型的变化,有可能增强病毒的结合能力。例如,SARS-CoV RBD区域的第487号残基苏氨酸(Thr)是稳定SARS病毒与受体结合的一个关键的位点(图4)。XFEP的计算结果显示,把此氨基酸位点突变成丙氨酸后损失的结合自由能的数值约为1.1 kcal/mol。而在2019-nCoV中,Thr487的位置被天冬酰胺(Asn)所取代后,该位点对RBD片段亲和力的贡献反而显著提升,突变为丙氨酸 (Ala) 后将损失4.3 kcal/mol的结合自由能 。
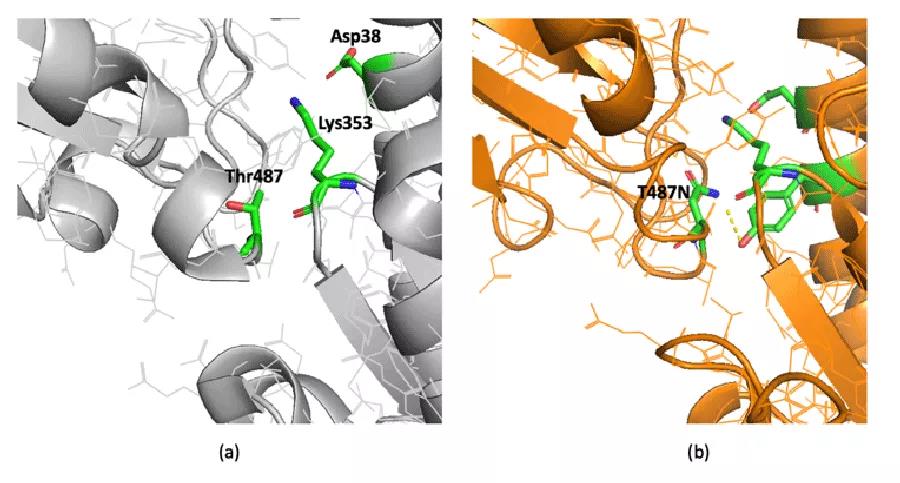
图 4:(a) 在SARS-CoV中,Thr487的甲基官能团指向ACE2结合界面上Lys353和Asp38形成的盐桥,起到稳定盐桥和结合界面的作用。(b) 在2019-nCoV中,Thr487位点上天冬酰胺的取代依然保留了对盐桥Lys353-Asp38的支撑,还与ACE2的Tyr41形成氢键,因而增强了复合物的亲和力和位点周围结构的稳定性。
另外一个例子是由SARS-CoV RBD 片段的第442号残基酪氨酸(Tyr)和第479号的天冬酰胺 (Asn) 所共同引起的局部相互作用的变化。在2019-nCoV中,这两个残基位点分别被亮氨酸 (Leu) 和谷氨酰胺 (Gln) 所取代( 图 5)。XFEP的扫描结果显示,N479Q和Y442L这两处突变均起到了稳定结合界面的作用:将442Tyr和479Asn突变为Ala损失的结合自由能的数值分别为1.1和0.8 kcal/mol, 而将Leu和Gln突变为Ala损失的结合自由能则更多,分别为3.9和1.3 kcal/mol。
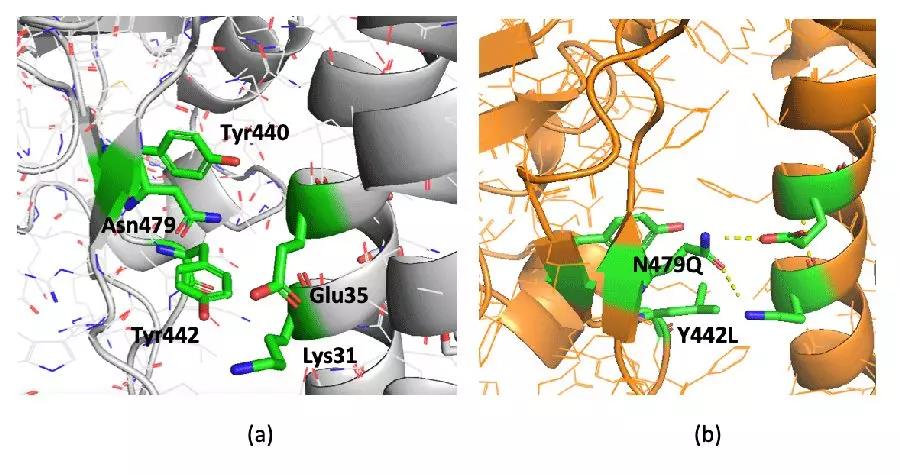
图 5:(a) 在SARS-CoV RBD和ACE2的结合界面上,Asn479被保护在附近的Tyr442和Tyr440形成的一个小的疏水口袋里,而对面的ACE2受体蛋白上的Lys31和Glu35通过电荷相互作用形成了盐桥的结构。(b) 2019-nCoV RBD-ACE2中, 替代Asn原有位置的Gln从疏水口袋中伸出,干扰了Lys31和Glu35之间的静电作用,并在整体构象调整的基础上,和两者分别形成了氢键相互作用。
当然,要判断新型冠状病毒与受体整体的结合能力,需要综合考虑结合界面上所有重要残基的贡献才能下结论。2019-nCoV属于RNA病毒,传播时S蛋白会较快发生突变【3】。如果能够提前获知哪些氨基酸的突变可以增强病毒的传染性,就可以提前开发相应的多肽或抗体类药物,快速对突变毒株做出响应,减少潜在的风险和损失。使用高精度的计算方法,可以准确评价氨基酸突变给病毒结合受体的能力带来的影响,并由此预测病毒的基因序列突变可能会给病毒传播能力带来的变化。
2.3CLpro和PLpro 潜在靶点的三维结构
在冠状病毒的非结构蛋白中,3CLpro和PLpro是治疗2019-nCoV的潜在靶点。晶泰科技基于1月20日 NCBI披露的2019-nCoV基因序列进行序列比对,发现其与SARS-CoV的基因序列最为相似,同源性高达79.5%。通过同源建模,我们根据SARS-CoV的蛋白晶体结构获得2019-nCoV的3CLpro和PLpro的三维结构,其中2019-nCoV-3CLpro结构与1月26日公布的晶体结构(PDB ID:6LU7,【4】)高度重合(RMSD:1.23 Å)。
3.老药新用筛选与活性排序
基于3CLpro和PLpro同源建模获得的三维结构,晶泰科技在其新药虚拟筛选平台上,对2900多个FDA已获批药物分子和超过10000个中药成分分子进行了活性筛选,共得到183个对3CLpro和PLpro靶点有潜在抑制作用的候选分子,并对他们的活性进行预测排序。其中,有针对2019-nCoV-3CLpr的上市药41个,中药成分分子36个;针对2019-nCoV-PLpro的上市药55个,中药成分分子41个。这一候选药物表单与其他机构近期筛选出的老药分子有所重合。目前,晶泰科技正在运用更高精度方法计算获得更精确的活性排位。
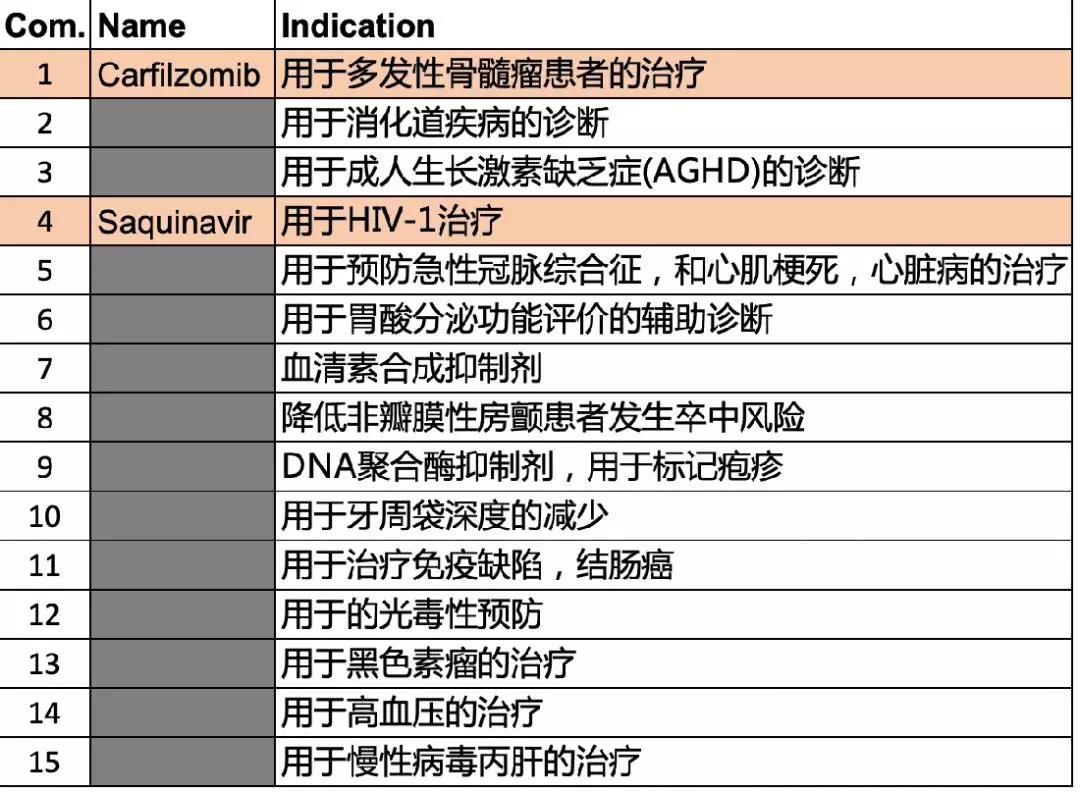
图6:对3CLpro靶点可能有抑制作用的活性排序靠前的已上市药物分子。其中,Carilzomib和Saquinavir与中科院上海药物所和上海科技大学联合研究团队发现的潜在药物列表重合
出于科学严谨性,暂仅对科研同行公开这一预测列表。欢迎具备病毒及分子层面快速实验验证能力的企业和研发机构与我们联系,共同加速有效治疗药物的筛选。
最后,感谢晶泰科技新冠状病毒研究小组的各位同事,在第一时间投入紧张的研发工作中,春节假期也不曾松懈。我们也要感谢坚守岗位的医务人员,感谢同样笔耕不辍、及时发布研究成果的科研同行,和所有在以自己的方式为抗击肺炎疫情贡献力量的人们。新型冠状病毒的创新疗法研发仍然任重而道远,但是绝不孤单。
参考资料:
1. Xu, X., Chen, P., Wang, J., Feng, J., Zhou, H., Li, X., Zhong, W. and Hao, P. (2020) Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission. Sci China Life Sci 63.
2. Li, F., Li, W., Farzan, M. and Harrison, S. (2005) Structure of SARS coronavirus spike receptor-binding domain complexed with receptor. Science 309, 1864-1868.
3. 已经出现了含有S蛋白突变的变异毒株,突变氨基酸为F32I https://nextstrain.org/ncov
4. 上科大和上海药物所联合研究团队预先公布新型肺炎冠状病毒3CL水解酶高分辨率晶体结构:http://siais.shanghaitech.edu.cn/2020/0126/c5404a50144/page.htm
本文标题图片修改自Public Domain
注:本文旨在介绍研究进展,不是治疗方案推荐。请患者前往正规医院就诊。
 664102020/02/11 15:00
664102020/02/11 15:00



 发布文章
发布文章 APP下载
APP下载







